유전자 발현
"오늘의AI위키"의 AI를 통해 더욱 풍부하고 폭넓은 지식 경험을 누리세요.
1. 개요
유전자 발현은 유전자의 DNA 염기 서열에 담긴 유전 정보를 바탕으로 생체 내에서 기능적인 RNA 또는 단백질이 생성되는 과정을 의미한다. 이 과정은 DNA의 전사, RNA의 형성, RNA의 세포질 투입, 운반 RNA의 작용, 단백질 형성, 단백질 접힘, 단백질 이동 등의 단계를 거쳐 이루어진다.
유전자 발현은 생물의 생존과 발달에 필수적이며, 발현 조절을 통해 세포는 환경 변화에 적응하고 다양한 기능을 수행한다. 유전자 발현 조절은 전사, 후전사 단계에서 이루어지며, 억제자, 활성자, RNA 간섭, 단백질 변형 등 다양한 기작이 관여한다. 또한, 유전자 발현은 발생 과정, 세포 분화, 생체 내 항상성 유지 등 생명 현상의 중요한 측면과 밀접하게 관련되어 있다.
유전자 발현은 바이러스 감염, 암, 항생제 내성 등 다양한 생물학적 현상을 이해하고 연구하는 데 중요한 지표가 되며, 전령 RNA, 단백질의 양을 측정하는 다양한 기술을 통해 유전자 발현 수준을 측정하고 분석할 수 있다.
더 읽어볼만한 페이지
2. 기제
유전자는 DNA의 특정 구간에 위치하며, 유전 형질의 단위가 된다. 유전자에 있는 유전 정보는 전령 RNA에 의해 전사되어 코돈 단위의 정보를 형성하고, 운반 RNA와 결합하여 아미노산 서열을 이룬 뒤 리보솜으로 운반되어 단백질을 생성한다.[147]
대장균을 모델로 한 실험을 통해 유전자 발현에 대한 많은 사실이 밝혀졌다. 대장균의 유전자 발현은 전사, 번역, 유전자 조절(발현 조절) 단계로 나눌 수 있으며, 주변 환경에 따라서도 조절을 받는다. 진핵생물의 유전자 발현 역시 기본적으로 진정세균과 동일하지만, RNA 중합효소의 종류가 더 많고, 전사 개시에 TATA 박스가 사용되는 등의 차이점이 있다.
2. 1. 전사
DNA의 염기서열이 RNA 중합효소에 의해 전령 RNA(mRNA)로 복사되는 과정이다. RNA 중합효소는 뉴클레오타이드 염기의 상보성 법칙에 따라 성장하는 RNA 가닥에 한 번에 하나의 리보뉴클레오티드를 추가한다. 이 RNA는 주형 3′ → 5′ DNA 가닥과 상보적이며, 티민(T)이 RNA에서는 우라실(U)로 대체된다는 점이 다르다.[1] 아데닌은 우라실과 짝을 이루고, 티민은 아데닌과, 구아닌은 시토신과 짝을 이룬다.[148]원핵생물은 한 종류의 RNA 중합효소만이 존재하여 전령 RNA, 운반 RNA, 리보솜 RNA를 모두 생산한다. 반면에 진핵생물은 각각의 RNA 종류에 따라 서로 다른 중합효소를 사용한다.[149]
세균에서 전사는 하나의 유형의 RNA 중합효소에 의해 수행되며, 전사를 시작하기 위해 시그마 인자 단백질(σ 인자)의 도움을 받아 프리브노 박스라고 하는 DNA 서열에 결합해야 한다. 진핵생물에서 전사는 핵에서 세 가지 유형의 RNA 중합효소에 의해 수행되며, 각 중합효소는 프로모터라고 하는 특수 DNA 서열과 일련의 DNA 결합 단백질—전사 인자—이 과정을 시작해야 한다.
- RNA 중합효소 I은 리보솜 RNA(rRNA) 유전자의 전사를 담당한다.
- RNA 중합효소 II(Pol II)는 모든 단백질 코딩 유전자뿐만 아니라 일부 비코딩 RNA(예: snRNA, snoRNA 또는 긴 비코딩 RNA)를 전사한다.
- RNA 중합효소 III은 5S rRNA, 전령 RNA(tRNA) 유전자 및 일부 작은 비코딩 RNA(예: 7SK)를 전사한다.
전사는 중합효소가 종결자라고 하는 서열을 만날 때 끝난다.
진정세균은 핵막을 가지고 있지 않으며, 유전자 전사의 장소와 번역의 장소가 진핵생물처럼 분리되어 있지 않다. 대장균의 게놈 DNA에서 전사가 진행 중인 mRNA는 신장 중에 5' 말단의 염기가 리보솜에서 번역되고 있다.
진핵생물의 유전자 발현 역시 기본적으로는 진정세균과 동일한 단계를 거치지만, 몇 가지 다른 점이 있다.
- RNA 중합효소의 종류가 많다(I, II, III이 존재하며, 유전자 인식 기구와 전사 유전자가 각각 다르다).
- 전사 개시에는 TATA 박스라는 티민, 아데닌이 풍부한 배열이 사용된다.
- 전사 종결 시스템은 불명확하며, 유전자의 2000염기쌍 하류까지 전사가 진행되기도 한다.
2. 2. RNA 형성
비부호화 RNA는 전단계의 형태로 존재하다가 필요한 과정에서 형성된다. 예를 들어 리보솜 RNA는 전리보솜 RNA로 전사된 뒤 핵소체에서 만들어지는 핵소체 저분자 RNA(SnoRNA)에 의해 절단되고 슈도우리딘 형태로 변형되어 형성된다.[150]운반 RNA의 경우 알엔에이스 피(RNase P)에 의해 5' 서열이 제거되고,[151] 3' 말단은 티알엔에이스 지(tRNase Z) 효소에 의해 제거되어 형성된다.[152][153]
2. 3. RNA 투입
대부분의 진핵생물은 핵소체에서 RNA를 만든다. 이렇게 만들어진 RNA는 핵공을 통해 세포질에 투입되어 단백질 형성 과정에 참여한다.[154] 시냅스와 같이 특별한 작용을 하는 세포질에는 RNA가 직접 투입되기도 하는데, 시냅스에서는 "지프코드"라 불리는 특별한 단백질이 모터 단백질을 이루어 RNA를 끌어당긴다.[155]2. 4. 운반 RNA
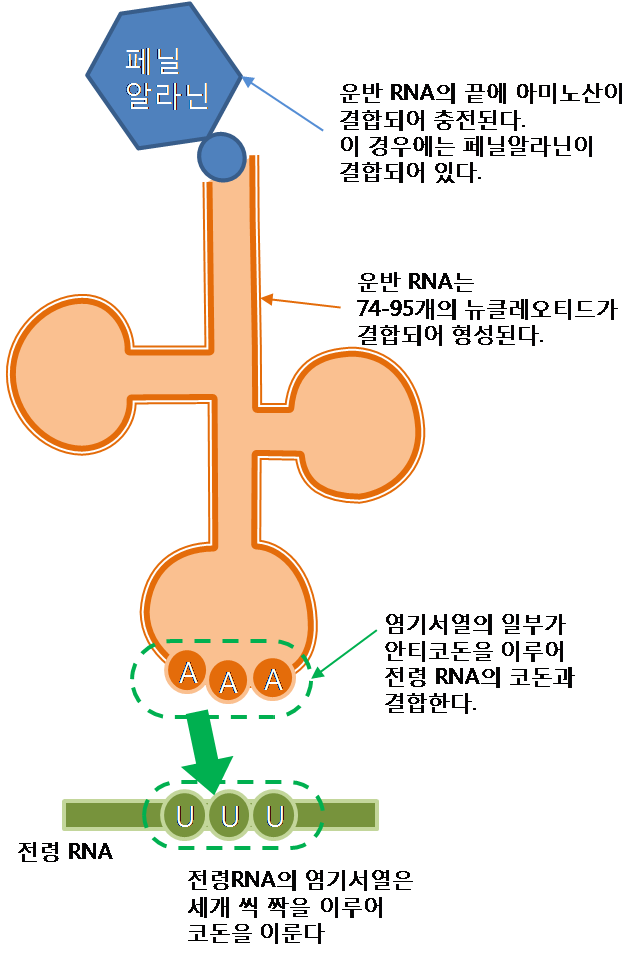
운반 RNA는 74 - 95 개의 뉴클레오타이드 염기 서열로 이루어진 저분자 RNA이다. 염기 서열의 끝에는 아미노산이 결합되고 염기 서열의 일부가 전령 RNA의 코돈과 상보적인 안티코돈을 이룬다.[156]
오른쪽 그림처럼 운반 RNA는 리보솜에서 전령RNA와 결합하고 리보솜은 운반 RNA에 연결된 아미노산을 떼어 내어 단백질 사슬에 연결한다.
운반 RNA에 의한 아미노산 운반 과정은 다음과 같다.[157]
- '''운반 RNA의 생성''': 운반 RNA의 염기서열 끝은 특정한 아미노산과 결합할 수 있는 구조로 되어 있다. 핵소체에서 20종의 모든 아미노산을 운반할 수 있는 운반 RNA가 생성된다.
- '''아미노산 충전''': 아미노아실 운반 RNA 합성 효소가 아미노산과 그에 맞는 운반 RNA를 결합시킨다.
- '''운반''': 아미노산이 충전된 운반 RNA는 리보솜으로 아미노산을 운반한다.
2. 5. 단백질의 형성

리보솜에서 전령 RNA(mRNA)의 코돈에 따라 아미노산이 결합하여 폴리펩타이드 사슬을 형성한다. 이 과정은 개시, 연장, 종료의 단계를 거친다.[158][159]
- '''준비'''
- 리보솜의 소단위체와 대단위체가 결합한다. 리보솜에는 운반 RNA가 들어설 수 있는 부위가 두 곳이 있는데, 운반 RNA가 들어오는 쪽을 A 자리, 나가는 쪽을 P 자리라 한다.
- '''초기화'''
- 소단위체에 전령 RNA가 결합하고, 개시 코돈이 확인되면 대단위체의 A 자리에 메티오닌과 결합한 운반 RNA가 도입되면서 단백질 생성이 시작된다.
- '''연장 과정'''
- '''코돈 인식''': 폴리펩타이드를 달고 있는 P 자리의 tRNA가 코돈에 붙어있다. A 자리에 있는 전령 RNA의 코돈과 상보성을 갖는 안티코돈을 가진 운반 RNA가 아미노산을 가져와 결합한다.
- '''펩타이드 결합 형성''': P 자리에 있는 기존의 폴리펩타이드와 A 자리에 있는 새로운 운반 RNA의 아미노산 사이에 펩타이드 결합이 형성된다. 이때 아미노아실 결합이 P 자리의 운반 RNA에서 새로운 폴리펩타이드 쪽으로 옮겨간다.
- '''변환''': P 자리에서 자유로워진 운반 RNA가 빠져나간다. 이제 전령 RNA가 한 칸 움직일 수 있게 되고 코돈이 바뀐다.
- '''반복''': A 자리의 폴리펩타이드-운반 RNA 중합분자가 P 자리로 옮겨가면서 A 자리가 비워진다. 비워진 A 자리에는 바뀐 코돈과 상보적인 운반 RNA가 새롭게 결합된다. 위 과정이 계속 반복되면서 폴리펩타이드 사슬이 길어진다.
- '''종료'''
- 종료 코돈에 종결 운반 RNA가 결합되면 합성되던 폴리펩타이드가 떨어져 나가면서 단백질 합성이 끝난다.
2. 6. 단백질 접힘
리보솜에서 생성된 폴리펩타이드는 1차원적인 아미노산의 사슬이다. 이렇게 늘어선 사슬은 기능에 맞게 3차원적으로 재구성되어야 하는데, 이 과정을 단백질 접힘이라 한다. 효소와 같은 단백질이 제 기능을 하기 위해서는 아미노산의 순서 뿐만 아니라 3차원 구조 역시 매우 중요하다.[160]각 단백질은 mRNA의 염기 서열로부터 아미노산의 선형 사슬로 번역될 때 펼쳐진 폴리펩타이드 또는 무작위 코일로 존재한다. 이 폴리펩타이드는 어떠한 발달된 3차원 구조도 갖지 않는다(그림의 왼쪽). 그런 다음 폴리펩타이드는 무작위 코일로부터 특징적이고 기능적인 3차원 구조로 접힌다.[30] 아미노산은 서로 상호 작용하여 잘 정의된 3차원 구조, 즉 접힌 단백질(그림의 오른쪽)을 생성하며, 이를 네이티브 상태라고 한다. 결과적인 3차원 구조는 아미노산 서열에 의해 결정된다(안핀센의 도그마).[31]
정확한 3차원 구조는 기능에 필수적이지만, 기능성 단백질의 일부는 접히지 않은 상태로 남아있을 수 있다.[32] 의도한 모양으로 접히지 못하면 일반적으로 독성 프리온을 포함하여 다른 특성을 가진 비활성 단백질이 생성된다. 여러 신경퇴행성 및 기타 질병은 '잘못 접힌' 단백질의 축적으로 인해 발생하는 것으로 여겨진다.[33] 많은 알레르기는 단백질의 접힘에 의해 유발되는데, 이는 면역계가 특정 단백질 구조에 대한 항체를 생성하지 않기 때문이다.[34]
샤페론이라고 불리는 효소는 새로 형성된 단백질이 기능을 수행하는 데 필요한 3차원 구조로 접히도록 돕는다.[35] 마찬가지로 RNA 샤페론은 RNA가 기능적 형태를 갖도록 돕는다.[36] 단백질 접힘을 돕는 것은 진핵생물의 소포체의 주요 역할 중 하나이다.
2. 7. 단백질 이동
세포 내에서 형성되는 단백질은 세포소기관을 형성하거나 효소를 구성한다. 또한 일부 단백질은 몸의 다른 곳에서 쓰이기 위해 이동한다. 소화 효소, 호르몬, 세포외기질과 같은 단백질들은 세포 밖에서 활동하기 위해 분비된다. 세포 내에서 만들어진 단백질의 분비는 골지체에서 조정된다.[161][162]진핵생물 또는 원핵생물의 분비 단백질은 분비 경로에 진입하기 위해 이동해야 한다. 새로 합성된 단백질은 신호 펩타이드에 의해 진핵생물 Sec61 또는 원핵생물 SecYEG 이동 채널로 이동한다. 진핵생물의 단백질 분비 효율은 사용된 신호 펩타이드에 매우 의존적이다.[37]
많은 단백질은 세포질이 아닌 다른 세포 부분으로 이동하며, 단백질이 가야 할 곳으로 안내하기 위해 다양한 신호 서열 또는 신호 펩타이드가 사용된다.[38][39] 원핵생물에서는 세포의 구획화가 제한적이므로 일반적으로 간단한 과정이다.[40] 그러나 진핵생물에서는 단백질이 올바른 소기관에 도달하도록 하기 위해 다양한 표적화 과정이 존재한다.[87]
모든 단백질이 세포 내에 남아 있는 것은 아니며, 많은 단백질이 수출된다. 예를 들어, 소화 효소, 호르몬, 세포 외 기질 단백질 등이 있다. 진핵생물에서 수출 경로는 잘 발달되어 있으며, 이러한 단백질의 수출을 위한 주요 메커니즘은 소포체로의 이동이며, 그 다음 골지체를 통한 수송이 이루어진다.[41][42]
3. 발현의 조절
생물 개체는 각각의 세포마다 유전체 전체가 들어 있지만, 유전자 발현은 필요한 부분에서 필요한 만큼만 일어나도록 조절된다.[163] 유전자 발현 조절은 유전자의 기능적 산물의 양과 출현 시기를 제어하는 것이다. 발현 조절은 세포가 필요할 때 필요한 유전자 산물을 생산할 수 있도록 하는 데 매우 중요하며, 이는 세포가 가변적인 환경, 외부 신호, 세포 손상 및 기타 자극에 적응할 수 있는 유연성을 제공한다. 더 일반적으로 유전자 조절은 세포가 모든 구조와 기능을 제어할 수 있도록 하며, 이는 세포 분화, 형태 형성, 그리고 모든 유기체의 다양성과 적응성의 기초가 된다.
얼룩무늬 고양이의 반점 색상은 피부의 여러 부위에서 생물학적 색소 유전자의 발현 수준이 다르기 때문에 나타난다.
유전자 발현 조절 방식에 따라 유전자 유형을 설명하는 데 사용되는 다양한 용어가 있다.
- '''구성 유전자'''는 선택 유전자와 달리 지속적으로 전사되는 유전자이다.
- '''하우스키핑 유전자'''는 기본적인 세포 기능을 유지하는 데 필요한 유전자이므로 일반적으로 유기체의 모든 세포 유형에서 발현된다. 예시로는 액틴, GAPDH, 유비퀴틴 등이 있다. 일부 하우스키핑 유전자는 비교적 일정한 속도로 전사되며, 이러한 유전자는 다른 유전자의 발현 속도를 측정하는 실험의 기준점으로 사용될 수 있다.
- '''선택 유전자'''는 구성 유전자와 달리 필요할 때만 전사되는 유전자이다.
- '''유도 유전자'''는 환경 변화에 반응하거나 세포 주기의 위치에 따라 발현이 달라지는 유전자이다.
유전자 발현의 모든 단계는 DNA-RNA 전사 단계에서 단백질의 번역 후 변형까지 조절될 수 있다. 최종 유전자 산물(RNA 또는 단백질)의 안정성 또한 유전자의 발현 수준에 영향을 미치는데, 불안정한 산물은 낮은 발현 수준을 초래한다. 일반적으로 유전자 발현은 DNA 전사[45] 및 RNA 번역[46]에 집합적으로 영향을 미치는 분자 간의 상호작용의 수와 유형의 변화[43][44]를 통해 조절된다.
유전자 발현이 중요한 몇 가지 간단한 예시는 다음과 같다.
- 인슐린 발현 조절을 통해 혈당 조절 신호를 제공한다.
- 암컷 포유류에서 유전자 "과다 복용"을 방지하기 위한 X 염색체 불활성화.
- 사이클린 발현 수준은 진핵 세포의 세포 주기 진행을 조절한다.
진정세균에서의 유전자 발현 조절은 자코브와 모노의 연구 논문을 기초로 이해할 수 있다. 발현 조절에는 '''오페론'''이라는, 몇몇 유전자가 짧은 간격을 두고 게놈 내에 나열되어 있는 구조체가 깊이 관여하는데, 그 시발점이 된 대장균의 락토스(유당) 대사에 대해 설명한다.
3. 1. 전사 조절
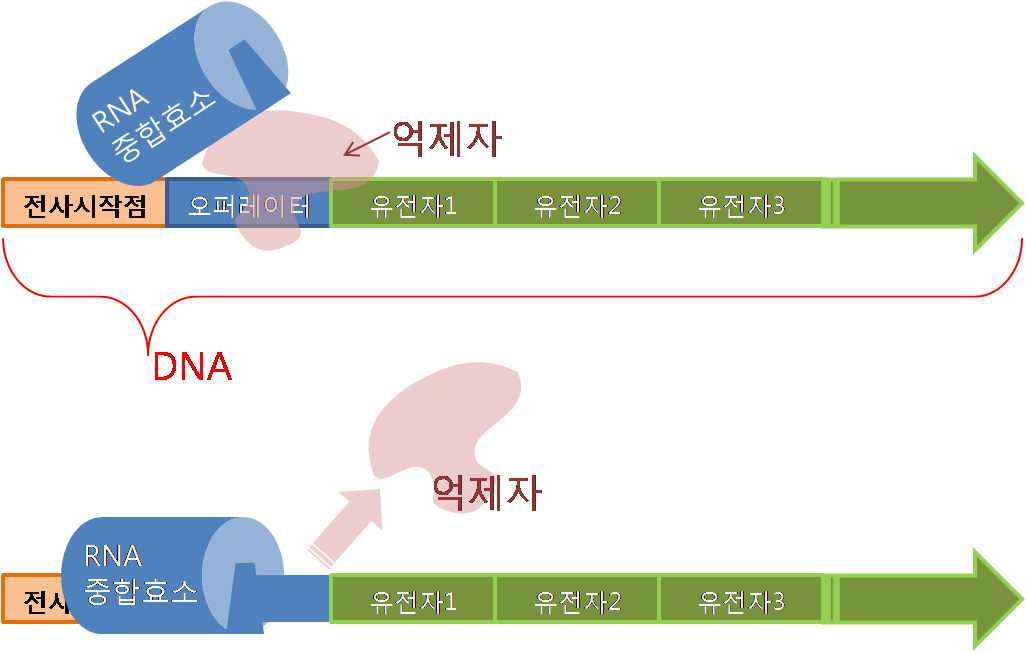
DNA를 전사하여 전령 RNA를 만드는 RNA 중합효소는 전사를 시작하는 DNA의 특정한 위치인 전사 개시점(프로모터)에 결합되어 전령 RNA를 만든다. 이 과정에서 억제자와 활성자 역할을 하는 효소가 전사를 조절한다.[164]- 억제자: 많은 유전자들은 필요한 때가 아니면 전사되지 않는다. 전사되지 않는 동안에는 억제자가 전사 시작점의 일부분인 오퍼레이터에 결합되어 있다. 이때문에 RNA 중합효소는 DNA가 열려 있어도 전사를 시작하지 못한다. 전사가 필요한 때에만 억제자가 DNA에서 분리되어 RNA 중합효소가 DNA와 결합되고 전령 RNA를 만들기 시작한다.
- 활성자: 한편, DNA에 RNA 중합효소가 결합하여 전사를 시작하기 위해서는 DNA의 이중나선이 열려야 한다. 활성자는 전사 시작점에 결합되어 DNA 이중나선을 풀고 RNA 중합효소가 결합하도록 돕는다.
- 조절: 세포 내에서 억제자와 활성자는 특정한 화학 성분의 변화에 민감하게 반응하여 조절된다. 예를 들어 락토스 당 신진대사를 하는 세균은 당분의 농도가 높아지면 억제자가 오페론에 부착되어 관련 소화 효소의 생산을 중단시킨다. 일정 시간이 지나 당분이 에너지로 소비되면 당분 농도가 낮아지고 억제자가 오페론에서 떨어져 나가 효소 생산이 다시 시작된다.

3. 2. 후전사 조절
RNA 중합효소의 작동을 조절하는 전사 조절과 달리 후전사 조절은 전사가 일어난 후에 여러 저분자 RNA가 간섭하여 단백질 형성을 조절하는 것이다. 특정 단백질 형성을 지시하는 전령 RNA(mRNA)가 리보솜에 가는 도중에 저분자 RNA에 의해 머리핀처럼 구부러져 상보적 뉴클레오타이드 사이에 접합이 일어나면, 이 전령 RNA는 더 이상 단백질 형성을 지시할 수 없게 된다. 이렇게 저분자 RNA에 의해 전령 RNA가 억제되는 것을 RNA 간섭이라고 한다. RNA 간섭은 강력한 유전자 발현 억제 기제로 작용한다.[165]진핵생물에서는 RNA의 번역이 가능하기 전에 RNA의 수출이 필요하며, 핵 수출은 유전자 발현에 대한 추가적인 제어를 제공하는 것으로 생각된다. 핵 안팎으로의 모든 수송은 핵공을 통해 이루어지며, 수송은 광범위한 임포틴 및 엑스포틴 단백질에 의해 제어된다.[86]
단백질을 암호화하는 유전자의 발현은 유전 암호를 전달하는 mRNA가 번역될 수 있을 만큼 오랫동안 생존하는 경우에만 가능하다.[87] 일반적인 세포에서 RNA 분자는 분해로부터 특별히 보호되는 경우에만 안정적이다.[88] RNA 분해는 mRNA가 번역되기 전에 상당한 거리를 이동해야 하는 진핵 세포에서 발현 조절에 특히 중요하다.[89] 진핵생물에서 RNA는 특정 전사 후 변형, 특히 5' 캡과 폴리아데닐화에 의해 안정화된다.[90]
mRNA의 의도적인 분해는 외래 RNA(일반적으로 바이러스)로부터의 방어 메커니즘뿐만 아니라 mRNA ''불안정화''의 경로로도 사용된다.[91] mRNA 분자가 작은 간섭 RNA에 상보적인 서열을 가지고 있다면, 이는 RNA 간섭 경로를 통해 파괴되도록 표적이 된다.[92]
3' 비번역 영역(3'UTR)은 mRNA의 유전자 발현에 전사 후 영향을 미치는 조절 서열을 종종 포함한다. 이러한 3'-UTR은 종종 마이크로 RNA(miRNA)와 조절 단백질의 결합 부위를 모두 포함한다.[93] miRNA는 3'-UTR 내의 특정 부위에 결합함으로써 번역을 억제하거나 전사체를 직접 분해하여 다양한 mRNA의 유전자 발현을 감소시킬 수 있다.[94] 3'-UTR에는 또한 mRNA의 발현을 억제하는 억제 단백질이 결합하는 사일런서 영역이 있을 수 있다.[95]
3'-UTR은 종종 마이크로 RNA 반응 요소 (MRE)를 포함한다. MRE는 miRNA가 결합하는 서열이다. 이것들은 3'-UTR 내에서 널리 퍼져 있는 모티프이다. 3'-UTR 내의 모든 조절 모티프(예: 사일런서 영역 포함) 중에서 MRE가 모티프의 약 절반을 차지한다.[96]
2014년 현재, miRBase 웹사이트[97]는 miRNA 서열 및 주석의 아카이브로서 233종의 생물종에 28,645개의 항목을 나열했다. 이 중 1,881개의 miRNA가 주석 처리된 인간 miRNA 유전자 좌에 있었다. miRNA는 평균 약 400개의 표적 mRNA (수백 개의 유전자 발현에 영향을 미침)를 가질 것으로 예측되었다.[98] 프리드먼(Friedman) 등[98]은 인간 mRNA 3'UTR 내에 45,000개 이상의 miRNA 표적 부위가 배경 수준 이상으로 보존되어 있으며, 인간 단백질 코딩 유전자의 60% 이상이 miRNA와의 쌍을 유지하기 위한 선택적 압력을 받아 왔다고 추정한다.
직접적인 실험 결과에 따르면, 단일 miRNA가 수백 개의 고유 mRNA의 안정성을 감소시킬 수 있다.[99] 다른 실험에서는 단일 miRNA가 수백 개의 단백질 생산을 억제할 수 있지만, 이러한 억제가 종종 비교적 경미하다는 것을 보여준다(2배 미만).[100][101]
miRNA의 유전자 발현 조절 장애의 영향은 암에서 중요한 것으로 보인다.[102] 예를 들어, 위장관 암에서 9개의 miRNA가 후성 유전학적으로 변경되어 DNA 복구 효소를 하향 조절하는 데 효과적인 것으로 확인되었다.[103]
miRNA의 유전자 발현 조절 장애의 영향은 정신분열증, 양극성 장애, 주요 우울증, 파킨슨병, 알츠하이머병, 자폐 스펙트럼 장애와 같은 신경 정신 질환에서도 중요한 것으로 보인다.[104][105]
3. 3. 발생과 유전자 조절
생물의 발생은 지속적인 유전자 조절의 과정이다. 하나의 수정란에서 시작되어 세포 분열과 증식, 그리고 특수한 기능을 가진 기관의 형성에 이르기까지 매 단계마다 유전자 발현은 지속적인 조절 과정을 거친다. 발생을 마친 개체의 각 세포는 최종적으로 분화된 역할을 일생 동안 계속한다. 예를 들어 양의 유방 세포는 젖 분비와 관련한 기능을 수행한다. 그러나, 특정한 역할만을 하는 체세포 역시 모든 유전체를 갖고 있기 때문에 조건만 맞는다면 다시 수정란과 같은 역할을 할 수 있다. 복제 생물 돌리는 이러한 체세포의 능력을 증명하는데, 정상적인 새끼를 낳아 생식 능력 역시 문제가 없음을 보였다.[166]4. 진핵생물과 원핵생물(세균)의 유전자 발현
원핵생물은 핵이 없어 DNA가 세포질에 존재하며, 전사와 번역이 거의 동시에 일어난다. 반면 진핵생물은 핵막으로 둘러싸인 핵 안에서 전사가 일어나고, 세포질에서 번역이 일어난다. 따라서 진핵생물의 전령 RNA(mRNA)는 핵에서 세포질로 이동해야 하며, 이 과정에서 여러 변형을 거친다.[14]
원핵생물의 mRNA는 전사 후 수정되지 않지만, 진핵생물의 mRNA는 5' 말단에 7-메틸구아노신 캡이 추가되고, 3' 말단에는 아데닌이 여러 개 붙는 폴리(A) 꼬리가 생성된다. 또한 RNA 스플라이싱 과정을 통해 인트론이 제거되고 엑손만 연결된다.[2]
리보솜의 크기도 차이가 있다. 원핵생물은 70S 리보솜을, 진핵생물은 80S 리보솜을 가진다. RNA 중합효소의 종류도 다른데, 원핵생물은 한 종류의 RNA 중합효소가 모든 종류의 RNA를 만드는 반면, 진핵생물은 RNA 종류에 따라 다른 중합효소를 사용한다.[149]
유전자 발현 조절 방식도 진핵생물이 원핵생물보다 복잡하며, 다양한 조절 인자가 관여한다.[1]
4. 1. 전사
유전자는 DNA의 일정 구간을 이루는 염기서열이다. 유전자의 구조는 전사의 시작과 끝을 알리는 엑손과 실제 전사가 진행되는 인트론 구간으로 이루어져 있다. 전사의 시작점에는 특수한 염기서열로 이루어진 프로모터가 있다. 프로모터는 RNA 중합효소가 DNA의 어느 지점에서 전사를 시작하여 어느 방향으로 진행할 것인지를 지시한다. 이 전사 개시점에서 이중나선 사슬이 풀리면서 전사가 시작된다. RNA 중합효소는 전사지시 구간의 오퍼레이터에 결합되어 DNA 사슬을 풀고 두 사슬 가운데 하나의 뉴클레오타이드와 상보성을 이루는 염기서열을 복제하여 전령 RNA를 만든다. 아데닌은 우라실과 짝을 이루고, 티민은 아데닌과, 구아닌은 시토신과 짝을 이룬다.[148] 만약 DNA의 염기 서열이 A-A-A 라면 RNA 중합효소가 만든 전령 RNA는 U-U-U 가 될 것이다.원핵생물은 한 종류의 RNA 중합효소만이 존재하여 전령 RNA, 운반 RNA, 리보솜 RNA를 모두 생산한다. 반면에 진핵생물은 각각의 RNA 종류에 따라 서로 다른 중합효소를 사용한다.[149]

DNA 가닥에서 RNA 사본을 생성하는 것을 전사라고 하며, RNA 중합효소에 의해 수행된다. RNA 중합효소는 뉴클레오티드 염기의 상보성 법칙에 따라 성장하는 RNA 가닥에 한 번에 하나의 리보뉴클레오티드를 추가한다. 이 RNA는 주형 3′ → 5′ DNA 가닥과 상보적이며,[1] 티민(T)이 RNA에서 우라실(U)로 대체된다는 점이 다르다.
세균에서 전사는 하나의 유형의 RNA 중합효소에 의해 수행되며, 전사를 시작하기 위해 시그마 인자 단백질(σ 인자)의 도움을 받아 프리브노 박스라고 하는 DNA 서열에 결합해야 한다. 진핵생물에서 전사는 핵에서 세 가지 유형의 RNA 중합효소에 의해 수행되며, 각 중합효소는 프로모터라고 하는 특수 DNA 서열과 일련의 DNA 결합 단백질—전사 인자—이 과정을 시작해야 한다. RNA 중합효소 I은 리보솜 RNA(rRNA) 유전자의 전사를 담당한다. RNA 중합효소 II(Pol II)는 모든 단백질 코딩 유전자뿐만 아니라 일부 비코딩 RNA(예: snRNA, snoRNA 또는 긴 비코딩 RNA)를 전사한다. RNA 중합효소 III은 5S rRNA, 운반 RNA(tRNA) 유전자 및 일부 작은 비코딩 RNA(예: 7SK)를 전사한다. 전사는 중합효소가 종결자라고 하는 서열을 만날 때 끝난다.
전사란 게놈 DNA에 코드된 유전자 본체 및 그 주변 영역이 RNA 중합 효소에 의해 상보적인 RNA 가닥(mRNA)으로 합성되는 과정이다. 필요한 재료로는 DNA 의존성 RNA 중합 효소(이하 RNA 중합 효소), 게놈 DNA, 리보뉴클레오티드가 기본적인 요소이다. 중합 효소의 반응 등에는 마그네슘 등을 요구하는 경우가 있지만, 여기서는 생략한다. 여기서 진핵생물과 다른 점은 '''RNA 중합 효소의 구조'''이다. 대장균의 RNA 중합 효소는 '''5개의 서브 유닛'''으로 구성되어 있으며, α2ββ’σ 구조(α 서브 유닛: 2개, β: 1개, β': 1개, σ: 1개)를 취하고 있다.
이러한 서브 유닛의 역할은 다음과 같다.
α는 '''무작위로''' 프로모터 서열에 결합하지만, σ 서브 유닛은 그 '''서열을 인식'''하여 발현에 적당한 유전자인지 판단한다. β 및 β'는 '''각각이 공역하여 RNA 중합 효소 활성을 발휘'''한다. σ 서브 유닛은 프로모터 서열 인식 시에 필요하며, 전사가 시작되면 RNA 중합 효소에서 떨어져 나간다. RNA 중합 효소에 포함되는지 여부에 따라 명칭이 다르며, 홀로 효소(α2ββ’σ 구조, 게놈 DNA의 프로모터 서열 인식 시에 구성되는 형상)와 코어 효소(α2ββ’ 구조, 전사 중, 그리고 세포 내를 유리하고 있는 상태의 형상)로 표시된다. σ 서브 유닛의 탈착은 전사 반응에 깊이 관련된다.
전사 반응은 개시, 신장(연장), 종결의 단계로 분류된다. 이러한 반응의 자세한 내용은 전사 항목에서 설명한다. 진핵생물의 유전자 발현 역시 기본적으로는 진정세균과 동일한 단계를 거치지만, 몇 가지 다른 점이 있다. 다만, 기본은 같으므로, 차이점을 언급하는 정도로 그친다.
- RNA 중합효소의 종류가 많다(I, II, III이 존재하며, 유전자 인식 기구와 전사 유전자가 각각 다르다).
- 전사 개시에는 TATA 박스라는 티민, 아데닌이 풍부한 배열이 사용된다.
- 전사 종결 시스템은 불명확하며, 유전자의 2000염기쌍 하류까지 전사가 진행되기도 한다.
4. 2. mRNA의 변형
진핵생물에서 mRNA는 여러 변형 과정을 거친다. 원핵생물에서는 전사와 번역이 거의 동시에 일어나며 mRNA가 수정되지 않는 반면, 진핵생물 유전자의 전사는 성숙한 RNA가 되기 위해 일련의 변형을 거쳐야 하는 RNA의 1차 전사체 (pre-RNA)를 생성한다.[2]pre-mRNA의 처리에는 다음 과정들이 포함된다.
- 5' 캡핑: pre-mRNA의 5' 말단에 7-메틸구아노신 (m7G)을 첨가한다. 이는 RNA를 엑소뉴클레아제에 의한 분해로부터 보호하는 효소 반응이다.[3] 캡 결합 복합체 이종이량체(CBP20/CBP80)는 m7G 캡에 결합하여 mRNA의 세포질 수출을 돕고, RNA를 디캡핑으로부터 보호한다.[4]
- 3' 절단 및 폴리아데닐화: 폴리아데닐화 신호 서열(5'- AAUAAA-3')이 pre-mRNA에 존재하면,[6] pre-mRNA는 먼저 절단된 다음 약 200개의 아데닌(A)이 첨가되어 폴리(A) 꼬리를 형성한다. 이는 RNA를 분해로부터 보호한다.[7] 폴리(A) 꼬리는 mRNA 수출 및 번역 재개시에 필요한 여러 폴리(A) 결합 단백질(PABPs)에 의해 결합된다.[8] CCR4-Not 3'-5' 엑소뉴클레아제는 디아데닐화 과정에서 폴리(A) 꼬리를 짧게 만들며, 이는 종종 전사체 붕괴로 이어진다.[9]
- RNA 스플라이싱: 대부분의 진핵생물 pre-mRNA는 엑손과 인트론으로 구성된다.[10] 스플라이소좀이라는 RNA-단백질 촉매 복합체는 스플라이싱 과정에서 두 개의 전이 에스테르화 반응을 촉매하여 인트론을 제거하고 라리아트 구조로 방출한 다음, 인접한 엑손을 함께 스플라이싱한다.[11] 경우에 따라 일부 인트론 또는 엑손을 성숙한 mRNA에서 제거하거나 유지할 수 있다.[12] 대체 스플라이싱은 단일 유전자에서 시작되는 일련의 서로 다른 전사체를 생성하며, 이는 잠재적으로 서로 다른 단백질로 번역될 수 있다. 따라서 스플라이싱은 진핵생물 유전자 발현의 복잡성과 종의 프로테옴 크기를 확장한다.[13]

- mRNA 편집: 드물게 전사 직후의 mRNA와는 전혀 다른 서열이 되는 경우도 있다.
원핵생물에서는 전사와 번역이 함께 일어나지만, 진핵생물에서는 핵막이 두 과정을 분리하여 RNA 처리가 발생할 시간을 제공한다.[14]
4. 3. 번역
번역은 전사된 mRNA의 코돈(유전 암호)에 따라, 리보솜에서 아미노산이 펩타이드 결합으로 중합체(단백질)가 되어가는 과정이다. 번역에 필요한 재료는 다음과 같다.- mRNA
- 아미노아실tRNA (20종류의 아미노산에 대응)
- 리보솜
진핵생물과 원핵생물은 리보솜의 크기가 다르다. 진정세균 및 고세균에서는 70S(Svedberg 단위), 진핵생물에서는 80S이다. 소서브유닛, 대서브유닛에 포함된 rRNA의 길이도 다르다(자세한 내용은 리보솜 참조).
번역 과정은 다음과 같은 단계로 분류된다.
- 개시
- 신장
- 종결
이러한 반응의 자세한 내용은 번역 (생물학) 항목에서 설명한다.
진정세균, 특히 대장균의 유전자 발현에서 전사와 번역은 거의 동시에 일어난다. 진정세균은 핵막이 없으며, '''유전자 전사의 장소와 번역의 장소가 진핵생물처럼 분리되어 있지 않다'''. 대장균 게놈 DNA에서 전사가 진행 중인 mRNA는 신장 중에 5' 말단의 염기가 리보솜에서 번역되고 있다. 진정세균의 mRNA는 수정되지 않기 때문에, 리보솜에서 합성된 폴리펩티드는 게놈 DNA의 유전자 배열 그대로의 아미노산 서열을 가지고 있다.
이 '''RNA 중합 효소와 리보솜의 공역 반응'''이야말로 진정세균에서의 유전자 발현의 실제라고 할 수 있다. 교과서 등에 게재된 유전 암호 표는 대장균을 기준으로 한 것이며 (정확히는, 대장균의 무세포 발현계를 사용), '''다른 생물이나 다른 유전자에서는 코돈과 아미노산의 대응이 다를 수도 있다'''. 예를 들어, 일반적으로 AGA는 아르기닌의 코돈이지만, 척추동물의 미토콘드리아에서는 종결 코돈으로 되어 있다.
진핵생물의 유전자 발현 역시 기본적으로는 진정세균과 동일한 단계를 거치지만, 몇 가지 다른 점이 있다.
- mRNA는 핵에서 세포질의 리보솜으로 운반되어 번역된다.
- 리보솜은 80S이다(60S+40S).
- mRNA에 샤인-달가노 염기서열(리보솜 결합 부위)이 없고, 캡 구조를 40S 리보솜이 인식한다.
- 포르밀메티오닌을 개시 코돈으로 사용하지 않는다(일반 메티오닌을 사용).
- 번역 개시에 ATP의 가수분해가 필요하다.
- 번역 종결에 GTP가 필요하다.
4. 4. 유전자 발현 조절
진핵생물의 유전자 발현 조절 기작은 원핵생물보다 복잡하며, 유전자 상류에 존재하는 다양한 발현 조절 인자가 관여한다. 예를 들어, 메탈로티오네인이라는 단백질 유전자의 경우, 한 줄의 상류에 9개의 조절 인자를 암호화하는 유전자가 알려져 있다.[1]TATA 박스, GC 박스, CAAT 박스, 인핸서, 사일렌서, 오퍼레이터, 억제자, 활성자, MRE 등 개별 유전자에 특유한 조절 인자도 많이 발견된다.[1]
5. 고세균의 유전자 발현
고세균의 유전자 발현은 진핵생물과 세균 양쪽의 특징을 모두 가지고 있다. 전사 양식은 진핵생물의 RNA 중합효소 II와 매우 유사하지만, 전사 후 mRNA의 수정은 일어나지 않는다. 번역 및 유전자 발현 조절도 중간적이다.
6. 히스톤의 아세틸화와 탈아세틸화
히스톤의 N-말단 라이신 잔기는 아세틸화, 탈아세틸화되며, 이는 유전자 발현 조절에 관여한다. 히스톤이 다수 아세틸화된 염색체 영역은 유전자의 전사가 활발하게 일어나며, 히스톤 아세틸화는 유전자 발현을 활성화시키고, 탈아세틸화는 유전자 발현을 억제하는 것으로 생각된다.[144][145]
7. 유전자 발현의 자기 조절
논코딩 RNA는 단백질을 사용하지 않고 전사 및 번역을 자기 조절할 수 있으며, 리보스위치는 이러한 mRNA에 포함되어 있는 조절 요소이다.
8. 유전자 발현 측정
생물학의 여러 분야에서 유전자 발현의 측정은 매우 중요하다. 예를 들어, 바이러스 감염 여부, 암 발병 가능성, 세균의 페니실린 내성 등을 확인할 수 있다. 유전자 발현을 측정하는 방법으로는 크게 전사된 mRNA를 측정하는 방법과 생성된 단백질을 측정하는 방법이 있다.[167][168]
유전자 발현 측정은 특정 유전자가 세포, 조직, 또는 유기체 내에서 얼마나 발현되는지를 수치화하여 생명 과학 분야에서 널리 활용된다. 이를 통해 다음과 같은 정보를 얻을 수 있다.
- 세포의 바이러스 감염 여부 (바이러스 단백질 발현)
- 개인의 암 발병 가능성 (종양 유전자 발현)
- 세균의 페니실린 내성 (베타-락타메이스 발현)
- 유전자 발현 프로파일링을 통한 세포 기작 이해 및 맞춤 의학에서 표적 치료 활용
단백질 발현 위치 분석은 유기체 또는 세포 수준에서 이루어지며, 발생 생물학 연구와 단일 세포 내 단백질 기능 연구에 중요하게 사용된다. 유전자 발현은 최종 산물인 단백질을 검출하여 측정하는 것이 이상적이지만, 보통은 mRNA를 검출하여 유전자 발현 수준을 추론하는 것이 더 간편하다.
8. 1. 전령 RNA 측정
유전체학에서는 고밀도로 집적된 올리고뉴클레오타이드인 마이크로어레이를 사용하여 전령 RNA(mRNA)의 농도를 측정한다. 이 올리고뉴클레오타이드들은 자신과 상보성을 이루는 전령 RNA와 결합한다. 올리고뉴클레오타이드는 인공적으로 합성하여 사용하는데 사진석판술을 이용하여 1.64 cm²의 유리판 위에 280,000개의 올리고뉴클레오타이드를 집적할 수 있다.[167]보다 진보된 기술로는 중합 효소 연쇄 반응(PCR)을 통한 유전자 발현 해석이 있다. 이는 전령 RNA를 기초로 인위적인 DNA를 역전사하는 방법이다. 전령 RNA의 코돈을 역전사 하면 원래의 DNA의 사슬이 되기 때문에 어떠한 유전자가 발현되었는지를 확인할 수 있다. 이렇게 전령 RNA를 기준으로 거꾸로 전사하여 만든 DNA를 상보성 DNA라 한다.[168]
mRNA의 수준은 mRNA 분자에 대한 크기 및 서열 정보를 제공하는 노던 블롯으로 정량적으로 측정할 수 있다.[114] RNA 샘플은 아가로스 겔에서 분리되고 표적 서열에 상보적인 방사성 표지 RNA 프로브와 혼성화된다.[115] 방사성 표지 RNA는 그 다음 오토라디오그래피에 의해 검출된다.[116] 방사성 시약을 사용하면 절차가 시간이 오래 걸리고 잠재적으로 위험하기 때문에, 디곡시제닌 및 바이오틴 화학과 같은 대체 표지 및 검출 방법이 개발되었다.[117] 노던 블롯의 단점은 대량의 RNA가 필요하고 겔 이미지에서 밴드 강도를 측정하는 것이 포함되어 있어 정량이 완전히 정확하지 않을 수 있다는 것이다.[118] 반면에, 노던 블롯으로부터 얻은 추가적인 mRNA 크기 정보는 대안적으로 스플라이싱된 전사체를 구별할 수 있게 해준다.[119][120]
mRNA 풍부도를 측정하는 또 다른 방법은 RT-qPCR이다. 이 기술에서는 역전사 후 정량적 PCR이 수행된다. 역전사는 먼저 mRNA로부터 DNA 주형을 생성한다; 이 단일 가닥 주형을 cDNA라고 한다. cDNA 주형은 다음 정량 단계에서 증폭되며, 이 과정에서 표지된 혼성화 프로브 또는 삽입 염료에서 방출되는 형광이 DNA 증폭 과정이 진행됨에 따라 변화한다.[121] 신중하게 구성된 표준 곡선을 사용하면 qPCR은 원래 mRNA 복제본의 절대적인 측정값을 생성할 수 있으며, 일반적으로 균질화된 조직의 나노리터당 복제본 수 또는 세포당 복제본 수 단위로 표시된다.[122] qPCR은 매우 민감하며 (단일 mRNA 분자 검출이 이론적으로 가능), 사용된 리포터 유형에 따라 비용이 많이 들 수 있다; 형광 표지된 올리고뉴클레오티드 프로브는 비특이적 삽입 형광 염료보다 비싸다.[123]
발현 프로파일링 또는 샘플 내의 많은 유전자에 대한 고처리량 분석의 경우, 저밀도 어레이의 경우 수백 개의 유전자에 대해 정량적 PCR을 동시에 수행할 수 있다.[124] 두 번째 방법은 혼성화 마이크로어레이이다. 단일 어레이 또는 "칩"은 하나 이상의 유기체의 게놈에 있는 모든 알려진 유전자의 전사체 수준을 결정하기 위한 프로브를 포함할 수 있다.[125] 또는, 유전자 발현의 직렬 분석(SAGE) 및 RNA-Seq와 같은 "태그 기반" 기술을 사용하여 서로 다른 mRNA의 세포 농도의 상대적 측정을 제공할 수 있다.[126] 태그 기반 방법의 장점은 "개방형 아키텍처"로, 알려지거나 알려지지 않은 서열을 가진 모든 전사체를 정확하게 측정할 수 있다는 것이다.[127] RNA-Seq와 같은 차세대 시퀀싱 (NGS)은 또 다른 방법으로, 참조 게놈에 매칭될 수 있는 방대한 양의 서열 데이터를 생성한다. NGS는 비교적 시간이 오래 걸리고 비용이 많이 들며 자원이 많이 필요하지만, 단일 염기 다형성, 스플라이스 변이체 및 새로운 유전자를 식별할 수 있으며, 서열 정보가 거의 또는 전혀 없는 유기체에서 발현을 프로파일링하는 데에도 사용할 수 있다.[128]
8. 2. 단백질 측정
웨스턴 블랏은 항체 형성 연구 등에서 이용되는 대표적인 단백질 직접 측정 방법이다.[169] 이 방법은 단백질의 크기와 정체성에 대한 정보를 제공한다. 샘플을 폴리아크릴아마이드 젤에서 분리하여 막으로 옮긴 후, 관심 있는 단백질에 대한 항체로 탐침한다. 항체는 이미징 및 정량화를 위해 형광체 또는 말무 효소에 접합될 수 있다. 젤 기반 분석법이라 정량화가 덜 정확하지만, 단백질 분해 또는 유비퀴틴화와 같은 단백질의 후기 변형 여부를 식별할 수 있다는 장점이 있다.[130]효소 결합 면역 흡착 분석법은 마이크로타이터 플레이트에 고정된 항체를 사용하여 시료에서 관심 단백질을 포획한다. 이후 효소 또는 형광체에 접합된 검출 항체를 사용하여 결합된 단백질의 양을 형광 측정법 또는 색도법으로 정확하게 측정할 수 있다. 이 방법은 젤 단계를 거치지 않아 더 정확한 정량화가 가능하다.
9. 유전자 발현 데이터베이스
참조
[1]
논문
Structure-function studies of the RNA polymerase II elongation complex
2009-02
[2]
서적
Lewin's genes XII
Jones & Bartlett Learning
2017-03-02
[3]
논문
mRNA capping: biological functions and applications
2016-09
[4]
논문
Cap-binding complex (CBC)
2014-01
[5]
논문
Cleavage and polyadenylation: Ending the message expands gene regulation
2017-07
[6]
논문
Transcripts synthesized by RNA polymerase III can be polyadenylated in an AAUAAA-dependent manner
2008-09
[7]
논문
Polyuridylation in Eukaryotes: A 3'-End Modification Regulating RNA Life
2015
[8]
논문
Roles of mRNA poly(A) tails in regulation of eukaryotic gene expression
2022-02
[9]
논문
CUCU modification of mRNA promotes decapping and transcript degradation in Aspergillus nidulans
2010-01
[10]
논문
Reflections on the history of pre-mRNA processing and highlights of current knowledge: a unified picture
2013-04
[11]
논문
RNAs in the spliceosome: Insight from cryoEM structures
2019-05
[12]
논문
Key features of the two-intron Saccharomyces cerevisiae gene SUS1 contribute to its alternative splicing
2011-10
[13]
논문
Alternative splicing as a regulator of development and tissue identity
2017-07
[14]
논문
On the origin of the nucleus: a hypothesis
2023-12
[15]
논문
Nucleolus: the fascinating nuclear body
2008-01
[16]
논문
Ribonuclease P: unity and diversity in a tRNA processing ribozyme
[17]
논문
tRNase Z
[18]
논문
tRNA maturation: RNA polymerization without a nucleic acid template
2004-10
[19]
논문
Functional diversity of small nucleolar RNAs
2020-02
[20]
논문
The Cajal body and histone locus body
2010-07
[21]
논문
Cajal body-specific small nuclear RNAs: a novel class of 2'-O-methylation and pseudouridylation guide RNAs
2002-06
[22]
논문
Exporting RNA from the nucleus to the cytoplasm
2007-10
[23]
논문
Cis-acting determinants of asymmetric, cytoplasmic RNA transport
2007-05
[24]
논문
The eukaryotic genome as an RNA machine
2008-03
[25]
논문
Maintenance of the correct open reading frame by the ribosome
2003-05
[26]
논문
Insights into protein biosynthesis from structures of bacterial ribosomes
2007-06
[27]
논문
Global quantification of mammalian gene expression control
http://edoc.mdc-berl[...]
2011-05
[28]
논문
Corrigendum: Global quantification of mammalian gene expression control
2013-03
[29]
논문
The concept of translocational regulation
2008-07
[30]
서적
Molecular Biology of the Cell
Garland Science
[31]
논문
The formation and stabilization of protein structure
1972-07
[32]
서적
Biochemistry
W. H. Freeman
[33]
논문
Folding proteins in fatal ways
2003-12
[34]
서적
Essential Cell Biology
Garland Science, Taylor and Francis Group, LLC
[35]
논문
In and out of the ER: protein folding, quality control, degradation, and related human diseases
https://works.bepres[...]
2007-10
[36]
논문
RNA misfolding and the action of chaperones
2008-01
[37]
논문
Optimized signal peptides for the development of high expressing CHO cell lines
2013-04
[38]
논문
Types of nuclear localization signals and mechanisms of protein import into the nucleus
2021-05
[39]
논문
Signal Peptide Features Determining the Substrate Specificities of Targeting and Translocation Components in Human ER Protein Import
2022-07-11
[40]
논문
Cell biology of prokaryotic organelles
2010-10
[41]
논문
The plant ER-Golgi interface: a highly structured and dynamic membrane complex
[42]
논문
Secretion without Golgi
2008-04
[43]
논문
Intranuclear trafficking: organization and assembly of regulatory machinery for combinatorial biological control
2004-10
[44]
논문
RNA regulation of epigenetic processes
2009-01
[45]
논문
The interplay between transcription factors and microRNAs in genome-scale regulatory networks
2009-04
[46]
논문
Regulation of mammalian gene expression by retroelements and non-coding tandem repeats
2008-04
[47]
논문
Transcriptional regulation and its misregulation in disease
2013-03
[48]
논문
The Role of the Ubiquitously Expressed Transcription Factor Sp1 in Tissue-specific Transcriptional Regulation and in Disease
2016-12
[49]
논문
Proteins Recognizing DNA: Structural Uniqueness and Versatility of DNA-Binding Domains in Stem Cell Transcription Factors
2017-08
[50]
논문
Understanding Transcription Factor Regulation by Integrating Gene Expression and DNase I Hypersensitive Sites
2015
[51]
논문
Enhancers and silencers: an integrated and simple model for their function
2012-01
[52]
논문
Defining mechanisms that regulate RNA polymerase II transcription in vivo
2009-09
[53]
논문
Regulation of transcription factor activity by interconnected post-translational modifications
2014-02
[54]
논문
Molecular genetics of the RNA polymerase II general transcriptional machinery
1998-06
[55]
논문
One thousand and one ways of making functionally similar transcriptional enhancers
2008-11
[56]
논문
The Nrf2-antioxidant response element signaling pathway and its activation by oxidative stress
2009-05
[57]
논문
Dysfunction of the ubiquitin-proteasome system in multiple disease conditions: therapeutic approaches
2008-11
[58]
논문
Switching Akt: from survival signaling to deadly response
2009-05
[59]
논문
Nonspecific transcription-factor-DNA binding influences nucleosome occupancy in yeast
2011-11
[60]
논문
Role of Epigenetics in Biology and Human Diseases
2016-11
[61]
서적
StatPearls
StatPearls Publishing
2024
[62]
서적
Epigenetics: Development and Disease
2013
[63]
논문
The Why of YY1: Mechanisms of Transcriptional Regulation by Yin Yang 1
2020
[64]
논문
Transcription factors: from enhancer binding to developmental control
2012-09
[65]
논문
Long-range enhancer-promoter contacts in gene expression control
2019-08
[66]
논문
YY1 Is a Structural Regulator of Enhancer-Promoter Loops
2017-12
[67]
논문
The Human Transcription Factors
2018-02
[68]
논문
Positional specificity of different transcription factor classes within enhancers
2018-07
[69]
논문
The Mediator complex: a central integrator of transcription
2015-03
[70]
논문
The degree of enhancer or promoter activity is reflected by the levels and directionality of eRNA transcription
2018-01
[71]
논문
MAP kinase phosphorylation-dependent activation of Elk-1 leads to activation of the co-activator p300
2003-01
[72]
논문
Enhancer RNAs predict enhancer-gene regulatory links and are critical for enhancer function in neuronal systems
2020-09
[73]
논문
DNA methylation in human epigenomes depends on local topology of CpG sites
2016-06
[74]
논문
Cytosine methylation and CpG, TpG (CpA) and TpA frequencies
2004-05
[75]
논문
Distribution, silencing potential and evolutionary impact of promoter DNA methylation in the human genome
2007-04
[76]
논문
Gene body methylation can alter gene expression and is a therapeutic target in cancer
2014-10
[77]
논문
Targeted DNA demethylation and activation of endogenous genes using programmable TALE-TET1 fusion proteins
2013-12
[78]
논문
Neural circuits and mechanisms involved in Pavlovian fear conditioning: a critical review
2006
[79]
논문
Experience-dependent epigenomic reorganization in the hippocampus
2017-07
[80]
논문
DNA Methylation and Establishing Memory
2022
[81]
논문
Primetime for Learning Genes
2017-02
[82]
논문
A genome-wide analysis of CpG dinucleotides in the human genome distinguishes two distinct classes of promoters
2006-01
[83]
논문
DNA methylation patterns and epigenetic memory
2002-01
[84]
논문
Cancer genome landscapes
2013-03
[85]
논문
MicroRNAs in the DNA Damage/Repair Network and Cancer
[86]
논문
The Great Escape: mRNA Export through the Nuclear Pore Complex
2021-10
[87]
서적
From RNA to Protein
https://www.ncbi.nlm[...]
Garland Science
2024-06-10
[88]
논문
mRNA stability in the nucleus
2014-05
[89]
논문
How do cells cope with RNA damage and its consequences?
2019-10
[90]
논문
Roles of mRNA poly(A) tails in regulation of eukaryotic gene expression
2022-02
[91]
논문
Mechanisms and consequences of mRNA destabilization during viral infections
2024-02
[92]
논문
microRNA inhibitors: Natural and artificial sequestration of microRNA
2017-10
[93]
논문
What Are 3' UTRs Doing?
2019-10
[94]
논문
Overview of MicroRNA Biogenesis, Mechanisms of Actions, and Circulation
2018-08-03
[95]
논문
Ciphers and Executioners: How 3'-Untranslated Regions Determine the Fate of Messenger RNAs
2019-01-24
[96]
논문
Frequency of MicroRNA Response Elements Identifies Pathologically Relevant Signaling Pathways in Triple-Negative Breast Cancer
2020-06
[97]
간행물
miRBase.org
[98]
논문
Most mammalian mRNAs are conserved targets of microRNAs
2009-01
[99]
논문
Microarray analysis shows that some microRNAs downregulate large numbers of target mRNAs
2005-02
[100]
논문
Widespread changes in protein synthesis induced by microRNAs
2008-09
[101]
논문
The impact of microRNAs on protein output
2008-09
[102]
논문
Mechanisms and role of microRNA deregulation in cancer onset and progression
2011-07
[103]
논문
Epigenetic reduction of DNA repair in progression to gastrointestinal cancer
2015-05
[104]
논문
The Emerging Role of microRNAs in Schizophrenia and Autism Spectrum Disorders
[105]
논문
MicroRNA and Posttranscriptional Dysregulation in Psychiatry
2015-08
[106]
논문
Regulation of translation initiation in eukaryotes: mechanisms and biological targets
2009-02
[107]
논문
The Variety in the Common Theme of Translation Inhibition by Type II Toxin-Antitoxin Systems
2020-04-17
[108]
논문
A Quick Guide to Small-Molecule Inhibitors of Eukaryotic Protein Synthesis
2020-11
[109]
논문
Protein posttranslational modifications: the chemistry of proteome diversifications
2005-12
[110]
논문
Proteome-wide post-translational modification statistics: frequency analysis and curation of the swiss-prot database
2011-09
[111]
논문
Proteomic analysis of post-translational modifications
2003-03
[112]
논문
Post-translational modifications and their biological functions: proteomic analysis and systematic approaches
2004-01
[113]
논문
Proteolytic post-translational modification of proteins: proteomic tools and methodology
2013-12
[114]
논문
A Comprehensive Analysis of Northern versus Liquid Hybridization Assays for mRNAs, Small RNAs, and miRNAs Using a Non-Radiolabeled Approach
2021-06
[115]
서적
Insect Molecular Genetics
https://www.scienced[...]
Academic Press
[116]
서적
Laboratory Methods in Enzymology: RNA
2013
[117]
논문
Labeling and colorimetric detection of nonisotopic probes
2001-05
[118]
논문
An overview of technical considerations when using quantitative real-time PCR analysis of gene expression in human exercise research
2018-05-10
[119]
논문
Modified Northern blot protocol for easy detection of mRNAs in total RNA using radiolabeled probes
2022-01
[120]
논문
Reduced stability of regularly spliced but not alternatively spliced p53 protein in PARP-deficient mouse fibroblasts
https://aacrjournals[...]
1999-01
[121]
논문
A PCR-based quantitative assay for the evaluation of mRNA integrity in rat samples
2018-05
[122]
서적
High-Throughput Next Generation Sequencing
https://link.springe[...]
Springer Science+Business Media
2011-03-22
[123]
논문
Low-cost SYBR Green-based RT-qPCR assay for detecting SARS-CoV-2 in an Indonesian setting using WHO-recommended primers
2022-11
[124]
논문
High throughput gene expression profiling: a molecular approach to integrative physiology
2004-01
[125]
논문
Overview of DNA microarrays: types, applications, and their future
2013-01
[126]
논문
Translating RNA sequencing into clinical diagnostics: opportunities and challenges
2016-05
[127]
논문
Methods for transcriptional profiling in plants. Be fruitful and replicate
2004-06
[128]
논문
RNA Sequencing and Analysis
2015-04
[129]
웹사이트
Search results < Expression Atlas < EMBL-EBI
https://www.ebi.ac.u[...]
[130]
논문
40 years Western blotting: A scientific birthday toast
2020-02
[131]
논문
The first comprehensive and quantitative analysis of human platelet protein composition allows the comparative analysis of structural and functional pathways
2012-10
[132]
논문
Poor transcript-protein correlation in the brain: negatively correlating gene products reveal neuronal polarity as a potential cause
2019-06
[133]
논문
Computational inference of gene regulatory networks: Approaches, limitations and opportunities
2017-01
[134]
논문
WebQTL: rapid exploratory analysis of gene expression and genetic networks for brain and behavior
2004-05
[135]
논문
Effects of hypoxia inducible factor-1alpha (HIF-1alpha) on the growth & adhesion in tongue squamous cell carcinoma cells
2009-02
[136]
논문
A combination of LongSAGE with Solexa sequencing is well suited to explore the depth and the complexity of transcriptome
2008-09
[137]
논문
The incredible shrinking world of DNA microarrays
2008-07
[138]
논문
High-resolution mapping of plasmid transcriptomes in different host bacteria
2009-01
[139]
논문
Annotating genomes with massive-scale RNA sequencing
2008
[140]
서적
Statistical Genomics
2016
[141]
논문
CollecTF: a database of experimentally validated transcription factor-binding sites in Bacteria
2014-01
[142]
논문
COLOMBOS v3.0: leveraging gene expression compendia for cross-species analyses
2016-01
[143]
논문
Many Microbe Microarrays Database: uniformly normalized Affymetrix compendia with structured experimental metadata
2008-01
[144]
웹사이트
アセチル化
http://www.cyclex.co[...]
null
2012-08-03
[145]
웹사이트
ヒストン脱アセチル化酵素(HDAC)阻害物質の分子設計とその抗がん剤への応用
http://pharm.life-bi[...]
null
2012-08-03
[146]
문서
Group of the Sequence Ontology consortium, coordinated by K. Eilbeck, cited in H. Pearson. (2006). Genetics: what is a gene? Nature, 441,
http://www.nature.co[...]
[147]
서적
분자생물학
기전연구사
2006
[148]
논문
Structure-function studies of the RNA polymerase II elongation complex
http://www.pubmedcen[...]
2009-02
[149]
서적
생명 생물의 과학
교보문고
2006
[150]
논문
Nucleolus: the fascinating nuclear body
http://www.pubmedcen[...]
2008-01
[151]
논문
Ribonuclease P: unity and diversity in a tRNA processing ribozyme
1998
[152]
논문
tRNase Z
2007
[153]
문서
RNA 사슬을 이루는 리보스는 다섯개의 탄소가 연결된 오탄당으로 3번과 5번 탄소의 연결을 통해 사슬을 이룬다. 따라서 사슬의 일정 구간을 끊기 위해서는 3번과 5번의 연결을 절단하는 효소가 필요하다.
[154]
논문
Exporting RNA from the nucleus to the cytoplasm
2007-10
[155]
논문
Cis-acting determinants of asymmetric, cytoplasmic RNA transport
http://www.ncbi.nlm.[...]
2007-05
[156]
웹사이트
Transfer RNA Structure
http://www.pdb.org/p[...]
[157]
서적
분자생물학
기전연구사
2006
[158]
서적
분자생물학
기전연구사
2006
[159]
서적
생명 과학
동화기술
2007
[160]
간행물
과학동아 2001년 9월호
동아사이언스
2001-09
[161]
논문
The plant ER-Golgi interface: a highly structured and dynamic membrane complex
2007
[162]
논문
Secretion without Golgi
http://www.pubmedcen[...]
2008-04
[163]
문서
이러한 유전자 발현 조절에 문제가 생길때 [[암]]이나 [[당뇨병]]과 같은 [[대사질환]]이 발생한다.
[164]
서적
생명 과학
동화기술
2007
[165]
서적
생명 과학
동화기술
2007
[166]
서적
생명 생물의 과학
교보문고
2006
[167]
서적
생물 공정 공학
교보문고
2003
[168]
서적
생화학
광문각
2009
[169]
웹사이트
Western blot antibody
http://www.exactanti[...]
2009-01-29
본 사이트는 AI가 위키백과와 뉴스 기사,정부 간행물,학술 논문등을 바탕으로 정보를 가공하여 제공하는 백과사전형 서비스입니다.
모든 문서는 AI에 의해 자동 생성되며, CC BY-SA 4.0 라이선스에 따라 이용할 수 있습니다.
하지만, 위키백과나 뉴스 기사 자체에 오류, 부정확한 정보, 또는 가짜 뉴스가 포함될 수 있으며, AI는 이러한 내용을 완벽하게 걸러내지 못할 수 있습니다.
따라서 제공되는 정보에 일부 오류나 편향이 있을 수 있으므로, 중요한 정보는 반드시 다른 출처를 통해 교차 검증하시기 바랍니다.
문의하기 : help@durumis.com

